Digitale Präzisionsgenomik revolutioniert die Pflanzenzüchtung: DiP-DIAMANT verbindet modernste Sequenzierungstechnologien mit Künstlicher Intelligenz für eine nachhaltige Bioökonomie.
Digitale Präzisionsgenomik für die Pflanzenzucht der Zukunft
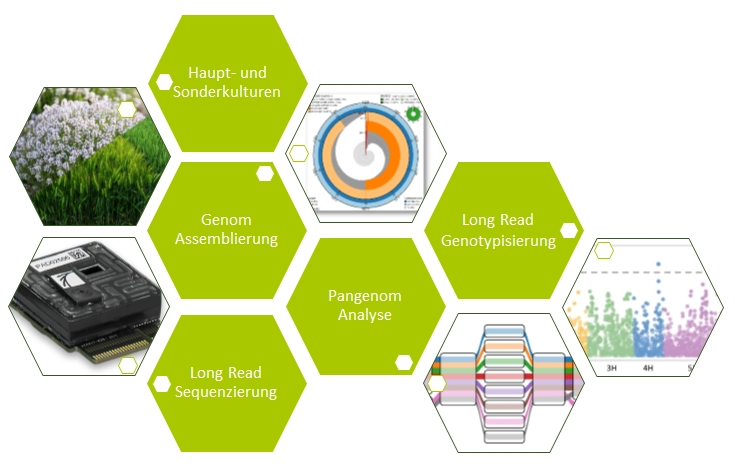
Die Züchtung klimaresistenter und ertragreicher Pflanzen erfordert hochpräzise genetische Analysen. DiP-DIAMANT nutzt Long-Read Sequenzierung und Künstliche Intelligenz, um verborgene genetische Potenziale sichtbar und gezielt nutzbar zu machen.
Am Institut für Agrar- und Ernährungswissenschaften der MLU Halle-Wittenberg entsteht eine digitale Plattform für Genomanalyse, die sowohl der Forschung als auch Züchtungsunternehmen völlig neue Möglichkeiten bietet. Besondere Schwerpunkte liegen auf Weizen und genetisch kaum erfassten Sonderkulturen für die neue Hochdurchsatz-Screening-Verfahren entwickelt werden. Diese Technologien ermöglichen eine schnellere, effizientere und nachhaltigere Pflanzenzucht.
DIP-DIAMANT stärkt damit nicht nur die Position Sachsen-Anhalts als Innovationsstandort für digitale Bioökonomie, sondern schafft auch konkrete wirtschaftliche Perspektiven. Die Verbindung von Spitzenforschung, digitaler Innovation und praktischer Anwendung macht die Region zu einem Vorreiter der modernen Pflanzenzüchtung.
• Entwicklung einer neuartigen Plattform für Long-Read-Genotypisierung und KI-basierte Genomanalyse
• Aufbau einer automatisierten Infrastruktur für Hochdurchsatz-DNA-Analysen
• Etablierung der Einzelzell-Technologie für komplexe Kulturpflanzen
• Erstellung digitaler Zwillinge von Pflanzensorten für präzisere Züchtung
• Stärkung der Region als Innovationszentrum für digitale Genomforschung
• Schaffung neuer Arbeitsplätze in der digitalen Bioökonomie
© 2025 DiP
Der beste Weg, um regelmäßig exklusive Einblicke und wertvolle Informationen zur Digitalisierung in der Landwirtschaft zu erhalten. (Erscheint demnächst 4x pro Jahr)